Projet
L’analyse d’image biologique occupe une place importante au sein du processus scientifique mais bien qu’il existe certains protocoles standardisés ces derniers manques de flexibilité et doivent être adapté au cas par cas aux différentes images.
Ces dernières années un nouvel outil d’analyse émerge rapidement, l’analyse par Deep-Learning. Ce dernier permet d’obtenir la flexibilité faisant jusque-là défaut.
En effet à partir du moment où un programme de Deep-Learning est entrainé avec un panel suffisamment varié d’images il est capable d’analyser une large gamme de ces dernières sans avoir besoin d’ajustement.
Certains programmes de Deep-Learning déjà entrainé ou permettant de faire un entrainement sans aucune connaissance en informatique nécessaire commencent à se démocratiser : Cellpose, Ilastick, Stardist.
Le RTmfm (Réseau Technologique de microscopie de fluorescence multidimensionnelle de la MITI du CNRS) a donc commencé (via son groupe de travail MAIIA) à créer des protocoles pour l’installation et l’utilisation de ces différents outils, ainsi qu’une réflexion au sens plus large sur le Deep-Learning.

Tracking
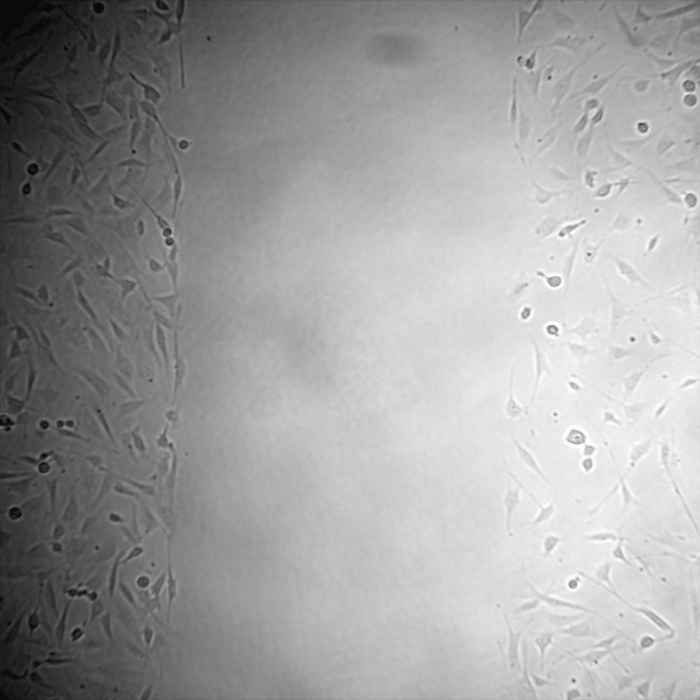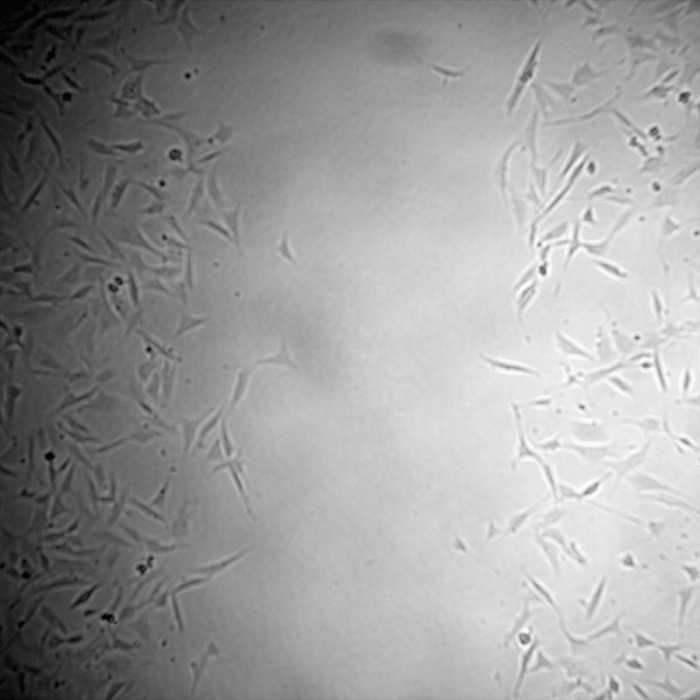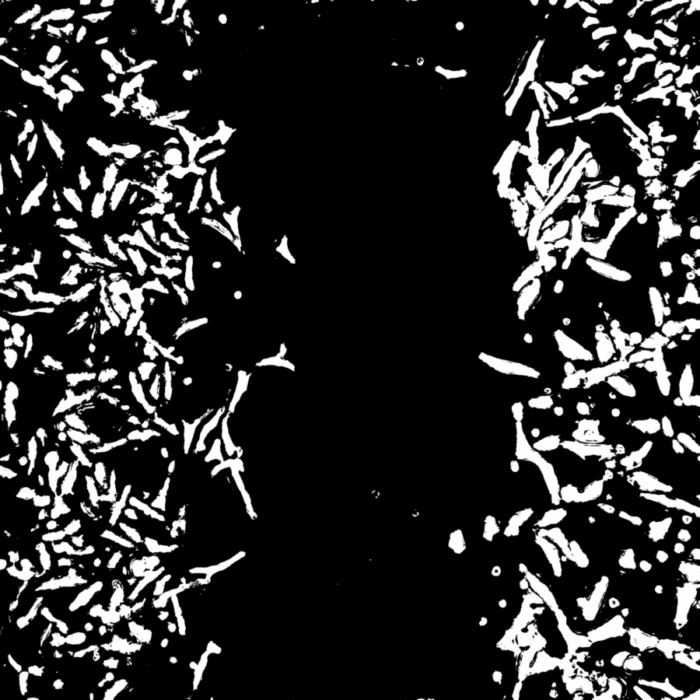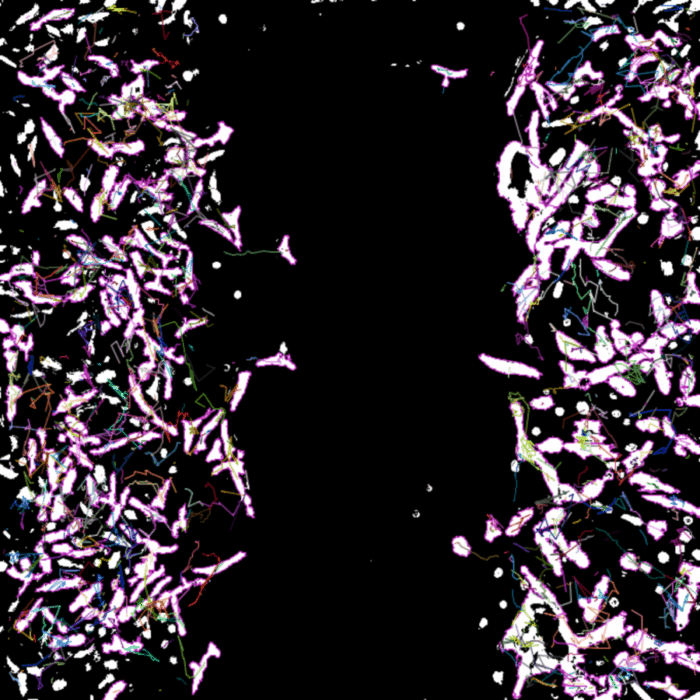
Exemple d’analyse de cellules acquises uniquement en lumière blanche sans contraste, avec une segmentation par le logiciel ilastik après apprentissage. Puis suivit des trajectoires de chaque cellule par le plugin TrackMate sur ImageJ